二十年前,当考虑更稳健蛋白质组学研究所需的工具探测细胞信号转导时,CST 的科学家提出一个创新性问题:如果抗体可能与翻译后修饰 (PTM) 物结合,而不考虑侧翼氨基酸序列,会怎么样?
这个问题和 CST 开发的所得方法就是 PTMScan® 系列产品和 CST® 蛋白质组学服务的基础,这些产品和服务通过允许研究人员使用液相色谱-串联质谱 (LC-MS/MS) 快速鉴定和量化数千个新颖 PTM 位点来简化蛋白质组学分析。
这篇文章探讨了如何产生这些革命性抗体以及使用该技术进行的一些开创性研究。
找到蛋白质组学研究的致命弱点
千禧年之交,CST 的科学家们正在努力克服许多研究人员面临的障碍。当时的 PTM 分析方法遭遇缺乏灵敏度、特异性和通量困扰,并且倾向于关注样品中更丰富存在的蛋白质。这可能意味着忽略了那些存在表达水平低但对疾病进展仍然至关重要的蛋白质的信号转导通路。
对于药物开发、突变分析和激酶分析研究,CST 科学家颠覆了传统的抗体开发过程并设计出一种至今仍然推动重要发现的全新 PTM 检测方法:PTMScan。
检测 PTM 的新方法:什么是 PTMScan?
PTMScan 使用独特的抗体开发方法,该方法使得在单次 LC-MS/MS 运行中检测数千个新 PTM 位点成为可能。该技术已成功用于监测磷酸化、泛素化、乙酰化和其他广泛研究的 PTM。
在创建 PTMScan 时,CST 科学家正在寻找一种在癌细胞中找出异常酪氨酸激酶活性的方法。1 激活的酪氨酸激酶是已知的致癌信号转导驱动因素,但鉴定特定底物可能有挑战性,原因是信号转导通路的复杂性和酪氨酸磷酸化水平低(<1% 的总磷酸化蛋白质组)。当时,可以对已知的酪氨酸磷酸化事件开发抗体,但如何鉴定新位点呢?
开发可检测新 PTM 位点的抗体
首先,让我们考虑一下科学家通常如何制造抗体。通常如此产生抗体:用肽抗原攻击兔的免疫系统以鉴定抗原特异性抗体。这产生位点特异性 PTM 抗体,这种抗体对靶标序列内部特定氨基酸残基处含 PTM 的单一肽序列或蛋白质具有亲和力。在下文例子中,使用单一磷酸肽作为抗原,并开发出仅对具有已限定磷酸酪氨酸残基的肽序列特异的抗体。
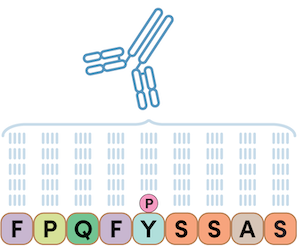
图 1. 当注入兔时,磷酸肽抗原可以产生磷酸化肽序列特异性抗体;在上文例子中,一种位点特异性磷酸酪氨酸抗体。PTMScan Direct 系列产品基于这项检测技术。
使用 PTMScan 时,不是开发单一磷酸肽序列特异性抗体,而是基于肽文库的方法用于抗体开发,这生成仅针对磷酸化氨基酸残基(或任何其他 PTM 残基)特异的抗体。该策略可适应于创造对其他翻译后修饰的残基(即乙酰赖氨酸、甲基精氨酸等)特异的抗体。在这种方法中,使用肽文库对兔免疫接种,以获得磷酸酪氨酸特异性抗体,其中磷酸酪氨酸位点固定并且所有其他侧翼位置都是随机化氨基酸1。
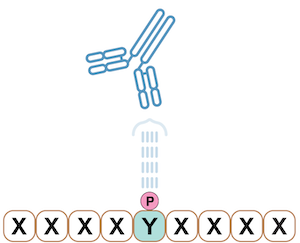 图 2:肽文库例子,其中磷酸酪氨酸位点固定,侧翼位置(标记为 X)是随机化氨基酸。上文示例显示了一种磷酸酪氨酸特异性抗体。这种技术用于创建 PTMScan Discovery 产品系列。
图 2:肽文库例子,其中磷酸酪氨酸位点固定,侧翼位置(标记为 X)是随机化氨基酸。上文示例显示了一种磷酸酪氨酸特异性抗体。这种技术用于创建 PTMScan Discovery 产品系列。
使用对任何磷酸酪氨酸位点特异的抗体,所有含磷酸化酪氨酸的肽都可以在消化后从复杂混合物中免疫沉淀出来,然后使用 LC-MS/MS 鉴定。
进一步扩展 PTM 特异性抗体方法以考虑磷酸化的底物基序。在这种方法中,使用肽文库对兔免疫接种,肽文库中磷酸丝氨酸位点和保守的底物基序固定,而其余的侧翼氨基酸随机化1。
下面的例子显示了一个基序抗体,其中精氨酸残基具有相对于磷酸丝氨酸为负三的固定位置。在肽文库中,这两个残基固定,而这些位置周围的氨基酸变动不定。只有丝氨酸残基磷酸化时,该基序抗体才会识别任何蛋白质中的序列 RXXpS,该序列为 AKT 通路信号转导中突出显示的 AKT 底物基序特有。
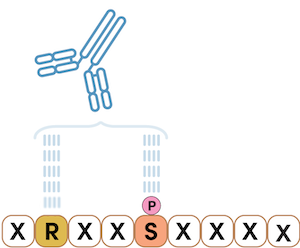
图 3. 仅固定两个位置就界定底物基序 RXXpS,其中 X 可以是任何氨基酸。在这个例子中,精氨酸残基具有相对于磷酸丝氨酸为负三的固定位置。基序抗体也用于 PTMScan Discovery。
使用这种方法,可以开发对任何靶向的修饰基序高度特异、而对多种不同一级氨基酸序列具有广泛反应性的抗体。
用于自下而上蛋白质组学的 CST 产品
使用上述抗体开发方法,CST 提供了一套 PTMScan 产品,这些产品有助简化用于靶向研究和初步发现工作的蛋白质组学分析,以高效专注于鉴定在您的模型系统内部驱动信号转导的关键 PTM。
PTMScan Direct
对于靶向研究,PTMScan Direct 在早期药物发现中鉴别中靶和脱靶,以帮助降低药物开发渠道风险。该技术允许对一组选定信号转导通路内部蛋白质靶标上的已知 PTM 进行以通路为中心的定向筛选,并可用于监测细胞对药物治疗的反应或鉴定特定疾病状态的驱动因素。PTMScan Direct 能够鉴定和量化信号转导通路中或细胞区室中从特定、代表性蛋白质衍生的数百个信号转导节点。
PTMScan Multi-Pathway Enrichment Kit 提供一种可靠的方法在单次实验中鉴定和定量剖析 >4000 个独特位点和 >800 种功能已知的关键蛋白质。
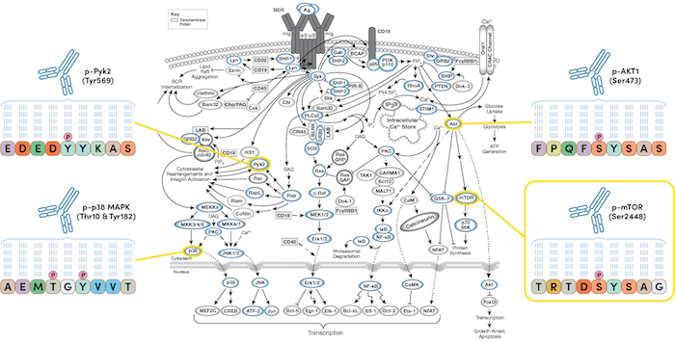
图 4. PTMScan Direct 可用于查询一组选定信号转导通路中蛋白质靶标上的已知 PTM。
PTMScan Discovery
用于发现模式研究的 PTMScan Discovery 搜索整个蛋白质组以鉴定和量化您的目标 PTM(图 5)。
使用 PTMScan Motif Antibody Kit 进行时,PTMScan Discovery 贯穿整个蛋白质组鉴别在对照状态和扰动状态之间受调节的关键 PTM,以揭示蛋白质上参与响应于测试条件或造成疾病进展(或表型)的关键位点。
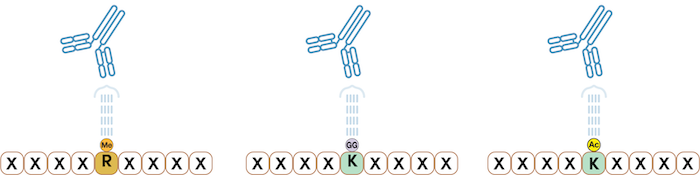 图 5. PTMScan Discovery 产品可以在您的样品中检出有意义的特定 PTM。左图:甲基精氨酸特异性抗体。中图:泛素/SUMO 残余物特异性抗体。右图:乙酰赖氨酸特异性抗体。
图 5. PTMScan Discovery 产品可以在您的样品中检出有意义的特定 PTM。左图:甲基精氨酸特异性抗体。中图:泛素/SUMO 残余物特异性抗体。右图:乙酰赖氨酸特异性抗体。
PTMScan HS:使用更少样品的蛋白质组学发现
为了提高 PTMScan 试剂的灵敏度和特异性,CST 科学家利用其专业知识来生产一种 PTMScan 抗体:磁珠偶联材料,这种材料导致酸洗脱期间释放自磁珠的抗体蛋白显著更少。这个新的 PTMScan 产品系列 (PTMScan HS for High Sensitivity) 还包括新的结合和洗涤缓冲液,以帮助减少背景肽结合。重要的是,这些试剂盒要求的样品更少,并且磁珠样式大大减少案头工作量并使其适于自动化。2
CST 蛋白质组学:PTMScan 服务
对于无法使用质谱设备但仍希望将发现研究或靶向 PTM 研究纳入其研究工作的研究人员,CST 蛋白质组学服务团队可以充当您实验室的延伸,使用上述技术定性和定量剖析您样品中的蛋白质或 PTM 位点。CST 科学家在整个项目期间为您提供咨询,从最初规划到交付一揽子数据包,确保您的研究目标实现。
使用 PTMScan 技术的发现成果
PTMScan 技术开发后,其迅速导致在肺癌3、卵巢癌4和急性髓性白血病5等疾病中发现新型酪氨酸磷酸化激活物。PTMScan 试剂盒的进一步商业化赋能全球实验室的科学家鉴定酪氨酸磷酸化的新位点及其上游激酶。现在,肿瘤学家掌握了将存在已知抑制物的患者与驱动增殖的激酶相匹配的信息。
CST 科学家很快开发出针对赖氨酸酰化(乙酰化、丁酰化、丙酰化和丙二酰化)的 PTMScan 抗体和鉴定蛋白质上泛素化位点和 SUMO 化位点的试剂。使用针对泛素残余物(即胰蛋白酶消化产生的基序)的独特抗体的 PTMScan 研究已经导致在数千种蛋白质上鉴定和发现数万个泛素化位点。此外,这些研究揭示了来那度胺治疗多发性骨髓瘤的作用机制6、多能性和细胞重编程的调节7、疱疹病毒侵袭表型中的泛素化需求8,以及关节炎中泛素连接酶的底物。9
最近,科学家们在研究黑色素瘤细胞中精氨酸甲基转移酶抑制剂的靶标时发现,mRNA 剪接因子的甲基化显著降低。10甲基转移酶抑制剂此前已被证明可以减缓某些癌细胞的生长。这些实验室采用旨在富集精氨酸甲基化位点的新型 PTMScan 试剂,并在癌细胞防卫中发现了另一个漏洞。这些剪切因子需要 PRMT5 对关键精氨酸残基进行甲基化,以正确剪切 mRNA。一些错误剪切的信息编码在调节 p53 和细胞周期中至关重要的蛋白质如 MDM4。这些实验以及使用 PTMScan 的类似实验已经鉴定对促进癌细胞生长关键的蛋白质。
PhosphoSite:PTM 在线数据库
在发明 PTMScan 的同时,CST 的科学家还创建了 PhosphoSitePlus.org,一个精选且在线可搜索的蛋白质磷酸化数据库。PhosphoSite 收录了已发表的蛋白质磷酸化数据以及我们内部研究的结果,从而形成了 PTM 在线数据库。11,12
此后,PhosphoSite 进一步扩展,囊括了蛋白质泛素化、乙酰化、赖氨酸甲基化、精氨酸甲基化和 N-糖基化,从而允许全球研究人员访问关于数十万个 PTM 的信息。
20 年革命性蛋白质组学研究
全球科学家已经使用 PTMScan 发表了 100 多篇经同行评审的稿件。PTMScan 的强大功能源自协同利用三项技术:PTM 特异性新抗体、尖端质谱和生物信息学。这种协同作用允许在一次实验中鉴定和相对量化数千个 PTM 位点。个体化医疗的前景取决于分子水平上确定异常生长的原因。二十年过去了,凭借无偏倚富集 PTM 肽的方法,PTMScan 技术仍然不失为一种表现出色的发现工具。
更多了解 CST 的蛋白质组学
- PTMScan 试剂盒可从 CST 网站购买,包括 PTMScan Pathway-Based Enrichment Kit 和 PTMScan Motif Antibody Kit。
- CST 还提供使用 PTMScan 的蛋白质组分析服务。
- 博客:3 倍数据,3 倍洞察力:DIA 和 DDA 蛋白质组学之间有什么区别?
- 博客:蛋白质组学实验中的可重复性:将对照肽与 PTMScan 结合使用
参考文献
- Rush J, Moritz A, Lee KA 等人Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nat Biotechnol. 2005;23(1):94-101. doi:10.1038/nbt1046
- Rivera KD, Olive ME, Bergstrom EJ, et al. Automating UbiFast for High-throughput and Multiplexed Ubiquitin Enrichment. Mol Cell Proteomics. 2021;20:100154. doi:10.1016/j.mcpro.2021.100154
- Rikova K, Guo A, Zeng Q, et al. Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer. Cell. 2007;131(6):1190-1203. doi:10.1016/j.cell.2007.11.025
- Ren H, Zhi-Ping T, Zhu X, Crosby K, Haack H, Ren JM, Beausoleil S, Mortiz A, Innocenti G, Rush J, Zhou X, Gu TL, Yang YF, Comb M. Identification of Anaplastic Lymphoma Kinase as a Potential Therapeutic Target in Ovarian Cancer. Cancer Res (2012) 72 (13) 3312-3323
- Gu TL, Goss VL, Reeves C 等人 Phosphotyrosine profiling identifies the KG-1 cell line as a model for the study of FGFR1 fusions in acute myeloid leukemia. Blood. 2006;108(13):4202-4204. doi:10.1182/blood-2006-06-026666
- Krönke J, Udeshi ND, Narla A 等人Lenalidomide causes selective degradation of IKZF1 and IKZF3 in multiple myeloma cells. Science. 2014;343(6168):301-305. doi:10.1126/science.1244851
- Buckley SM, Aranda-Orgilles B, Strikoudis A 等人Regulation of pluripotency and cellular reprogramming by the ubiquitin-proteasome system. Cell Stem Cell. 2012;11(6):783-798. doi:10.1016/j.stem.2012.09.011
- Huffmaster NJ, Sollars PJ, Richards AL, Pickard GE, Smith GA. Dynamic ubiquitination drives herpesvirus neuroinvasion. Proc Natl Acad Sci U S A. 2015;112(41):12818-12823. doi:10.1073/pnas.1512559112
- Lee KA, Hammerle LP, Andrews PS 等人Ubiquitin ligase substrate identification through quantitative proteomics at both the protein and peptide levels. J Biol Chem. 2011;286(48):41530-41538. doi:10.1074/jbc.M111.248856
- Musiani D, Bok J, Massignani E 等人Proteomics profiling of arginine methylation defines PRMT5 substrate specificity. Sci Signal. 2019;12(575):eaat8388. Published 2019 Apr 2. doi:10.1126/scisignal.aat8388
- Hornbeck PV, Zhang B, Murray B, Kornhauser JM, Latham V, Skrzypek E. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Res. 2015;43(Database issue):D512-D520. doi:10.1093/nar/gku1267
- Johnson JL, Yaron TM, Huntsman EM 等人 An atlas of substrate specificities for the human serine/threonine kinome. Nature. 2023;613(7945):759-766. doi:10.1038/s41586-022-05575-3
CST 和 Cell Signaling Technology 是 Cell Signaling Technology, Inc. 的注册商标。所有其他商标均为其各自所有者的财产。访问 cellsignal.com/trademarks 以了解更多信息。 23-BPA-73950
23-BPA-73950


 沪公网安备31011502018823号
沪公网安备31011502018823号