如果您已执行 CUT&RUN、CUT&Tag 或 ChIP/ChIP-seq 实验,您知道使用高质量抗体生成您可以信任的数据是多么重要。但是,您是否知道 CST 还提供将会确保数据质量和可重复性的染色质剖析测定法支持试剂?
本博客讨论非抗体支持试剂可能如何影响您的数据,并回答您在优化染色质剖析实验时可能遇到的以下问题:
- 我应该将哪些磁珠用于 ChIP/ChIP-seq 实验?
- 为 ChIP/ChIP-seq 超声处理染色质时,如何保护染色质和抗体表位?
- 以 MNase 片段化用于 ChIP/ChIP-seq 的染色质时,如何最大程度地减少变异性?
- 可以在 CUT&RUN 或 CUT&Tag 实验中使用 DNA 纯化柱吗?
- 我应将哪种洋地黄皂苷浓度用于 CUT&RUN 或 CUT&Tag 实验?
- 我应该在 CUT&RUN 和 CUT&Tag 实验中使用多少 pAG-MNase 和 pAG-Tn5?
CST 在此支持您的所有染色质剖析需求——开发、配制并严格验证这些辅助试剂,然后才到达您的工作台,从而确保您的数据质量。奖励?这些试剂可作为现成的 CUT&RUN、CUT&Tag 或 ChIP/ChIP-seq 试剂盒提供或以单件形式可提供,以方便您使用。
毕竟,谁会愿意为此花费那么多时间进行实验只是得到实验失败或产生模棱两可的结果,而原因在于缓冲液或酶未按应有方式发挥作用?
我应该将哪些磁珠用于 ChIP/ChIP-seq 实验?
蛋白 G 琼脂糖或磁珠与抗体联合使用,以富集染色质结合的目的蛋白。然而,有许多类型的磁珠可用。您如何知道哪些磁珠将提供最佳的染色质免疫沉淀 (ChIP) 数据和 ChIP-seq 数据?CST 开发了 ChIP 级磁珠,让您更轻松地选择磁珠。
CST 科学家评估了不同类型的珠,以确定那些提供最佳富集和最小背景者。在开发 ChIP 级磁珠时,还评估了不同的偶联化学。为了产生更好的信噪比,ChIP-Grade Protein G Magnetic Beads #9006 在含 BSA 的缓冲液中配制,以阻断非特异性蛋白结合,而 ChIP-Grade Protein G Agarose Beads #9007 在含 BSA(以阻断非特异性蛋白结合)和经超声处理的鲑精 DNA(以阻断非特异性 DNA 结合)的缓冲液中供应。
请注意,如果您正在进行 ChIP-seq,则将需要使用 ChIP 级磁珠,因为与琼脂糖珠结合的鲑精 DNA 可能污染您的测序结果。
与 IP 级磁珠相比,ChIP 级磁珠产生更好的信噪比

图 1. 利用 ChIP-Grade Protein G Magnetic Beads #9006 或 IP-Grade Protein A Magnetic Beads,使用来自 HeLa 细胞的已消化染色质和所示抗体进行 ChIP。使用针对所示基因的引物,通过实时定量 qPCR 分析纯化的 DNA。纯化 DNA 的表达为染色质总输入的百分比。
为 ChIP/ChIP-seq 超声处理染色质时,如何保护染色质和抗体表位?
超声处理是一种用于使染色质片段化,从而其可溶且轻易共沉淀的方法。然而,超声处理可能非常严苛且可能导致损坏染色质和抗体结合表位。将 CST 的细胞裂解缓冲液和胞核裂解缓冲液配制成更好地保护染色质和抗体表位的完整性,但仍有效使染色质段化并使抗体可用于免疫沉淀 (IP)。这在研究转录因子和辅助因子时尤其重要,因为它们与染色质的结合比组蛋白标志物更弱,而且往往丰度更低。
与竞品相比,SimpleChIP® Sonication Cell and Nuclear Lysis Buffers #81804,也作为 SimpleChIP Plus Sonication Chromatin IP Kit #56383 的一部分提供,对组蛋白标志物提供等同的下一代测序(NGS)结果。然而,转录因子和辅因子的 NGS 信噪比表明,与竞品相比,CST 缓冲液有助于提供优异结果。
在分析转录因子或辅因子相互作用时,CST 超声处理和胞核裂解缓冲液的性能优于竞品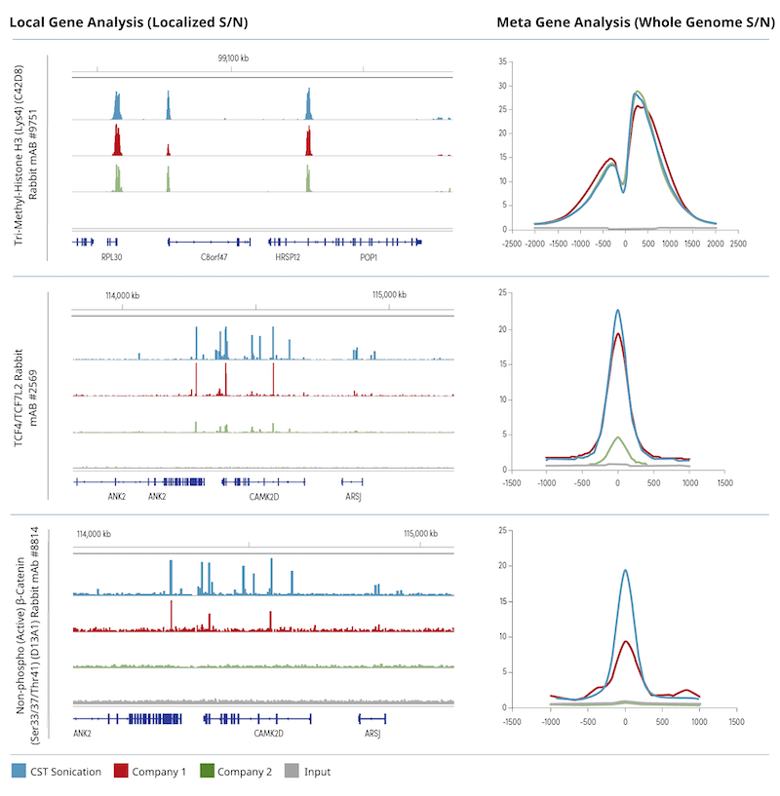
图 2. 根据试剂盒制造商提供的方案,使用来自 4 x 106 个HCT 116 细胞的交联染色质制备染色质,并使用所示的抗体进行免疫富集。左边的 ChIP-seq 迹线表明目的蛋白跨局限化目的区域富集。边的荟萃基因分析描述了跨全基因组中存在的所有已鉴定峰的信噪比。输入样本用作 ChIP-seq 迹线视图和迹线荟萃基因分析的阴性对照。
以 MNase 片段化用于 ChIP/ChIP-seq 的染色质时,如何可以最大程度减少变异性?
使染色质片段化时,另一个选择是使用微球菌核酸酶 (MNase) 而不是超声处理。使用 MNase 时需要考虑的是,即使按相同的浓度使用,酶活性也可能在批间变动。为了帮助您避开这种来源的分析间不一致性并节省您的时间,CST 在验证期间对 Micrococcal Nuclease #10011 的酶活性(而不是浓度)进行标准化。对每个新批次的 MNase 进行滴定实验,以比照之前批次平行评估活性,因此向四百万个细胞添加 0.5 µL 来自 CST 的 MNase 时,您始终会得到相同的染色质消化样式。
在下面的示例中,配制新批次的 MNase 以显示与既往批次相同的活性,两者均产生所需的染色质消化样式。
依据酶活性使 MNase 标准化确保了批间一致性
图 3. 将四百万个细胞/消化进行甲醛交联,并且使用渐增量的两个不同批次 Micrococcal Nuclease #10011 ,如 SimpleChIP® Plus 染色质免疫沉淀实验步骤(磁珠)中所述制备并消化染色质。消化的染色质 DNA 样品在 1% 琼脂糖凝胶上如所示分离。泳道 4 和泳道 9 中显示所需的染色质消化带样式。
可以在 CUT&RUN 或 CUT&Tag 实验中使用 DNA 纯化柱吗?
CUT&RUN 和 CUT&Tag 是替代 ChIP-seq 的一种更快且更具成本效益的方法。开发 CUT&RUN 测定法和 CUT&Tag 测定法的 Henikoff 实验室最初建议使用苯酚-氯仿提取纯化 DNA,因为 CUT&RUN 生成的 DNA 片段比 ChIP 生成的片段小。遗憾的是,苯酚-氯仿是一种非常混乱且耗时的方法。纯化柱更易用,但并非所有 DNA 纯化柱都同等地造出。CST 的科学家已经验证,我们的纯化柱高效纯化 CUT&RUN 测定法和 CUT&Tag 测定法中生成的较小 DNA 片段。
DNA Purification Buffer and Spin Columns (ChIP, CUT&RUN, CUT&Tag) #14209 回收 ≥ 35 bps 的 DNA 片段,确保回收超过 98% 的 CUT&RUN DNA 片段,因为这些片段中不到 2% 者小于 35 bps。这些柱还与 CUT&Tag 兼容,因为标签化片段至少为 70 bps,因为接头在纯化前已连接到目的 DNA。每个新批次柱的 DNA 片段回收率均依据真实染色质剖析试验来确认,这些试验保证为您的首选应用提供一致性能。
用于 CUT&RUN 片段的 DNA 纯化方法比较。
图 4.(A) 将低量程 DNA 标准品混合物(泳道 1,未纯化)使用 DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN, CUT&Tag) #14209(泳道 2)或苯酚/氯仿提取继而乙醇沉淀法(泳道 3)纯化,并通过在 4% 琼脂糖凝胶上电泳来分离。如所示,苯酚/氯仿提取继而乙醇沉淀法高效回收所有大小的 DNA 片段,而 DNA 离心柱法回收 ≥ 35 bp 的 DNA 片段。(B) 在使用 TCF4/TCF7L2 (C48H11) Rabbit mAb #2569 的 CUT&RUN 测定法中使用苯酚/氯仿提取继而乙醇沉淀法纯化 DNA。使用 Bioanalyzer 系统分析文库中 DNA 片段的大小。构建期间向文库添加的接头和条码序列占了 140 bp 片段长度。因此,起始的 35 bp DNA 片段长度将在文库制备后变为 175 bp(图中用蓝色垂直线标注)。如所示,CUT&RUN 富集的总 DNA 片段中不到 2% 短于 175 bp(起始长度 35 bp),这提示 DNA 纯化离心柱足以捕获 > 98% 的总 CUT&RUN DNA 片段。
我应将哪种洋地黄皂苷浓度用于 CUT&RUN 或 CUT&Tag 实验?
进行 CUT&RUN 和 CUT&Tag 实验时使用的洋地黄苷需要使细胞和核膜透化到刚好足够到酶和抗体可以进入,但到每种物质(包括 DNA )可以出来的程度,因为这会增加分析背景。如同 MNase,即便使用相同的洋地黄苷浓度,洋地黄苷活性也可能逐批变动。因此,应进行滴定实验评估活性。
CST 会滴定每个新批次的 Digitonin Solution #16359 ,以确保批间活性水平相同,从而为您节省时间。每个新批次都与之前批次平行受测并根据信号强度而不是浓度进行标准化。然后使用 NGS 比照既往批次,测试新批次的最佳浓度,以确认性能同等。
洋地黄皂苷滴定确保批次间活性一致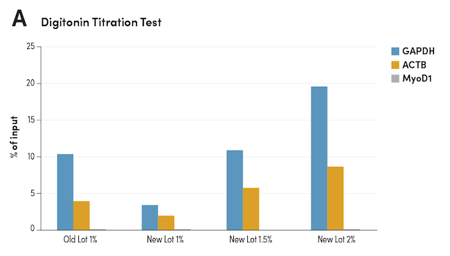

图 5. 使用 CUT&RUN Assay Kit #86652,以 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit #9751 进行 CUT&RUN。(A)如所指示在缓冲液中使用不同批次具有不同浓度的 Digitonin Solution #16359。使用人 GAPDH 外显子 1 引物、SimpleChIP ® Human β-Actin Promoter Primers #13653 和 SimpleChIP ® Human MyoD1 Exon 1 Primers #4490,通过实时 PCR 对富集的 DNA 定量。每个样品中富集 DNA 的量表示为相对于输入 DNA 总量的信号。(B)在缓冲液中使用新批次 Digitonin Solution #16359 的最佳浓度 1.5%,并使用 NGS 与既往批次平行比较。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。该图显示结合作用遍及 GAPDH,GAPDH 是 H3K4me3 的已知靶标基因。
我应该将多少 pAG-MNase 和 pAG-Tn5 用于 CUT&RUN 和 CUT&Tag 实验?
CST 已针对 CUT&RUN 和 CUT&Tag 分别开发并配制了 pAG-MNase 酶和 pAG-Tn5 酶,以便性能和批次间可重现性最佳。对每个新批次验证过酶活性而不是浓度,以确保结果一致。
涉及使用的量时,用于进行 CUT&RUN 实验的 pAG-MNase 酶有耐受性。然而,在配制和验证附属于 CUT&RUN Assay Kit #86652 以及 CUT&RUN pAG-MNase and Spike-In DNA #40366 的 pAG-MNase 时,CST 仍会比照之前既往批次进行平行滴定和 NGS 测试。
依据酶活性使 pAG-MNase 标准化确保了批间一致性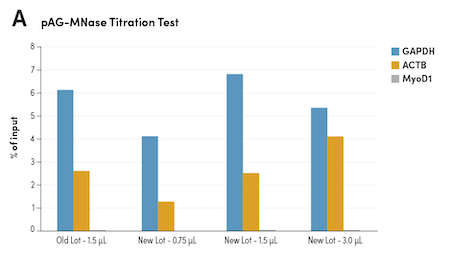

图 6. 使用 CUT&RUN Assay Kit #86652,以 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit #9751 进行 CUT&RUN。(A)如所指示在缓冲液中使用不同批次具有不同浓度的 pAG-MNase #40366。使用人 GAPDH 外显子 1 引物、SimpleChIP ® Human β-Actin Promoter Primers #13653 和 SimpleChIP® Human MyoD1 Exon 1 Primers #4490,通过实时 PCR 对富集的 DNA 定量。每个样品中富集 DNA 的量表示为相对于输入 DNA 总量的信号。(B)测定法中使用最佳体积 1.5 μl的 新批次 pAG-MNase #40366,并使用 NGS 与既往批次进行平行比较。使用 DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 制备 DNA 文库。该图显示结合作用遍及 GAPDH,GAPDH 是 H3K4me3 的已知靶标基因。
成功的 CUT&Tag 测定法更依赖于 pAG-Tn5 酶的活性而不是使用的数量,因为在我们的内部测试中,每个反应中使用超过推荐量的酶时,并未观察到读段数目、峰数目或信噪比增加(在 CUT&Tag 常见问题解答页面中了解更多详情)。为了确保批次间性能一致,用 NGS 通过将新批次与既往批次平行滴定来验证 pAG-Tn5 (Loaded) #79561 酶(还随附 CUT&Tag Assay Kit #77552)。pAG-Tn5 酶的活性会随着时间的推移而减弱,因此必须将酶储存在 -20°C 下,并在小瓶上列出的有效期之前使用酶。
通过酶活性将 pAG-Tn5 标准化可确保批间一致性
图 7. 使用 CUT&Tag Assay Kit #77552,以 HCT 116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 进行 CUT&Tag。按照所示,在测定中使用不同批次且不同量的 CUT&Tag pAG-Tn5 (Loaded) #79561。使用 CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415 制备 DNA 文库。该图显示结合作用遍及 GAPDH,GAPDH 是一种已知的 H3K4me3 靶标基因。
博客:CUT&Tag DNA 文库产率:如果使用 Agilent Bioanalyzer 或 TapeStation 系统时您的文库产量太低以致于检不出,怎么办
CST:您的一站式染色质分析商店
使用 CST 开发和验证的 ChIP、CUT&RUN 和 CUT&Tag 试剂,不仅可以节省您的时间,而且可以减少麻烦。我们的科学家花费了大量时间优化缓冲液、酶和 DNA 纯化柱,以便您获得最佳结果。我们还为您进行滴定,并将其与之前的批次进行比较,以确保批间一致性,从而保证随着时间推移的可重复性。再加上针对您选择的应用而经过严格验证的抗体,您将在不知不觉中生成您认可的染色质分析数据。
23-FLE-89700


 沪公网安备31011502018823号
沪公网安备31011502018823号